This is the Linux app named FOCUS whose latest release can be downloaded as FOCUS_0.31_python_2.7.XX.zip. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named FOCUS with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start the OnWorks Linux online or Windows online emulator or MACOS online emulator from this website.
- 5. From the OnWorks Linux OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application, install it and run it.
SCREENSHOTS
Ad
FOCUS
DESCRIPTION
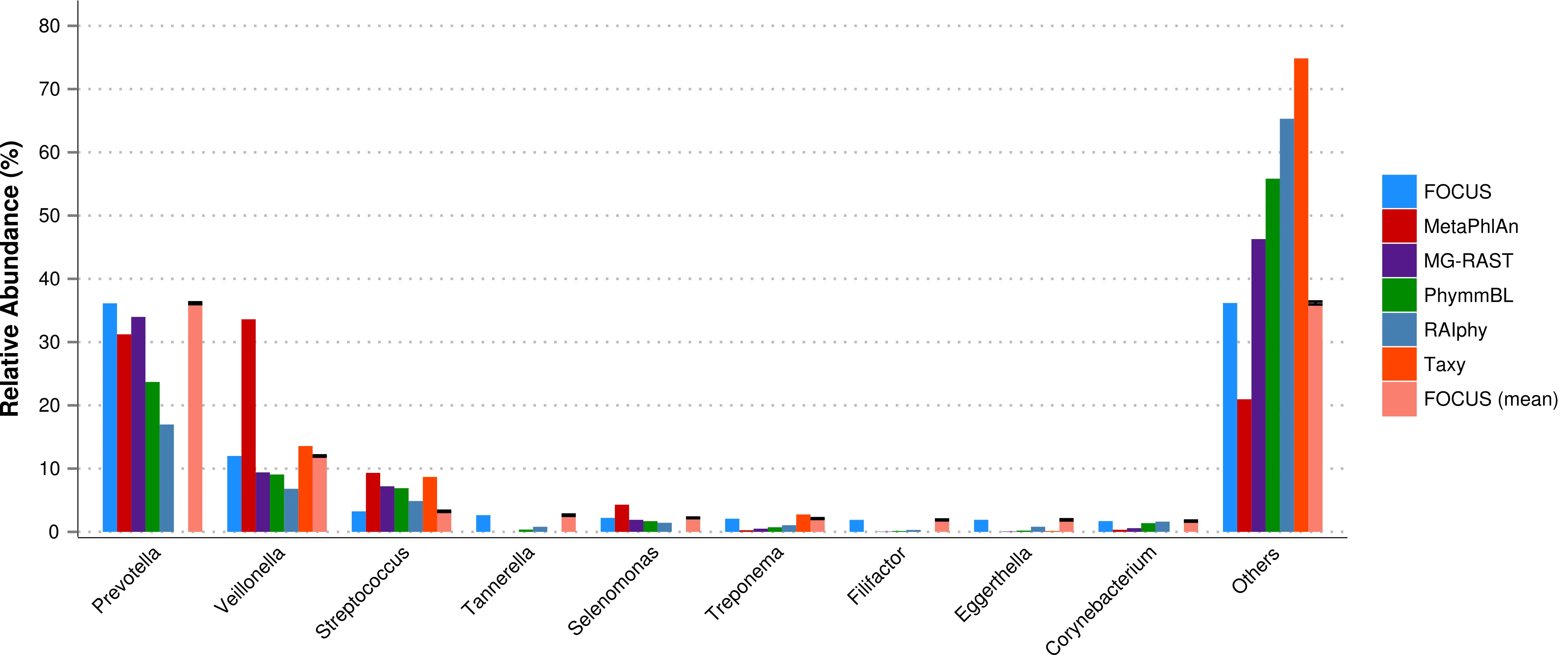
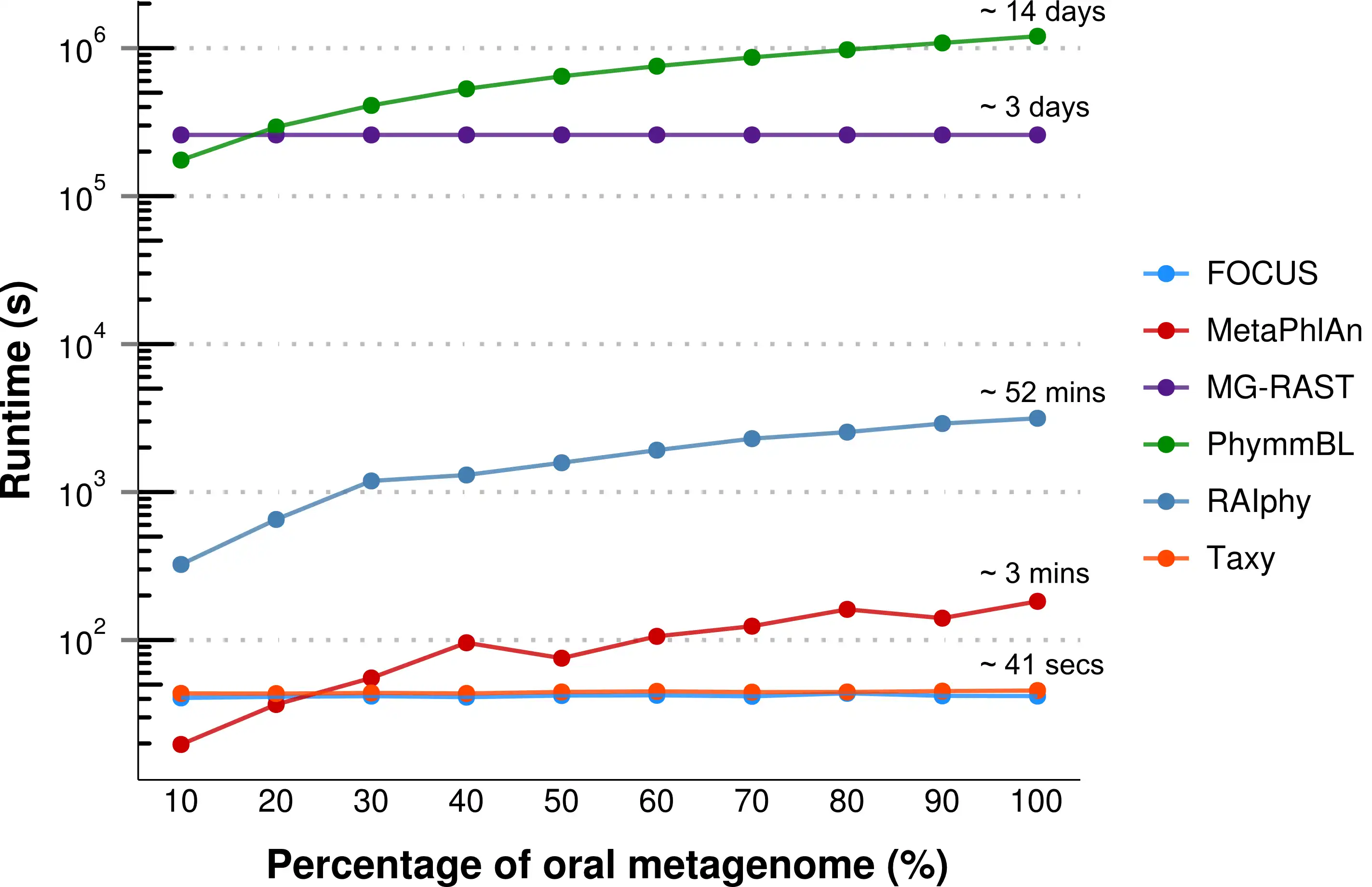
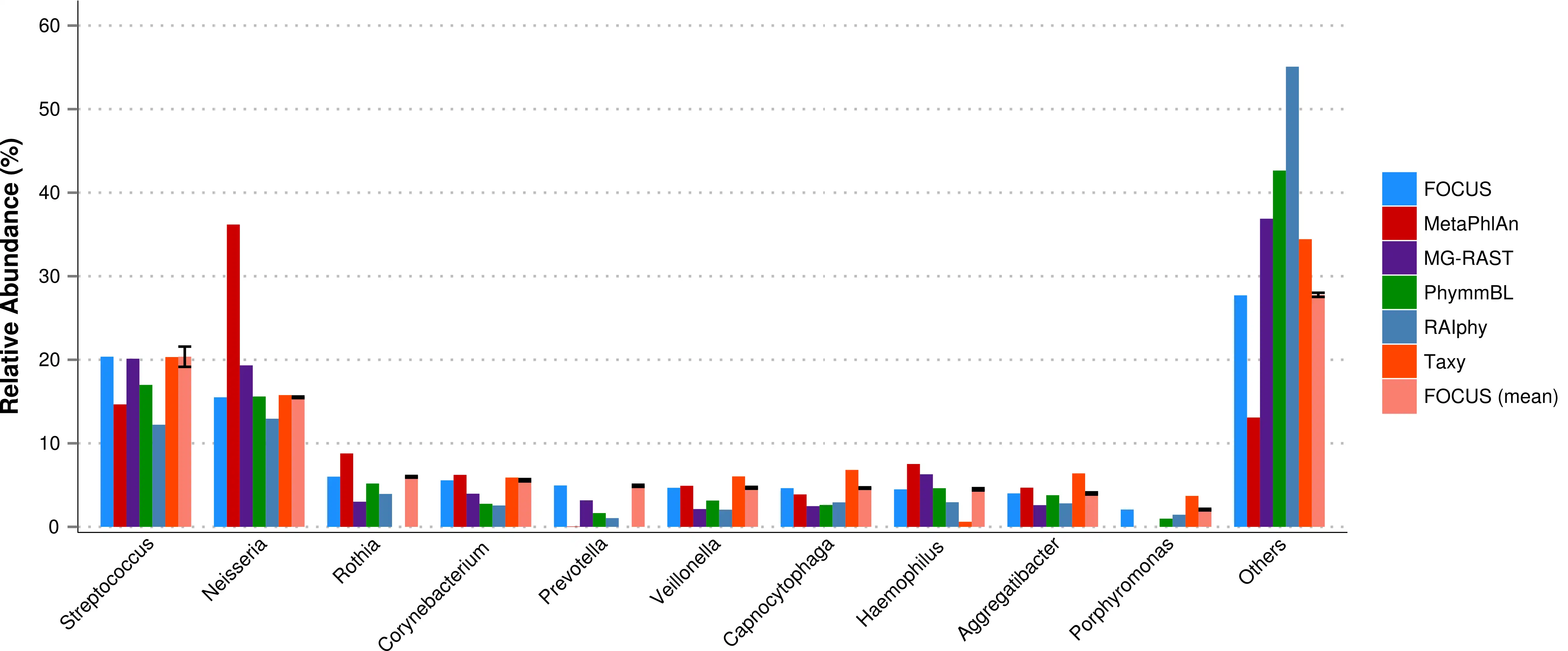
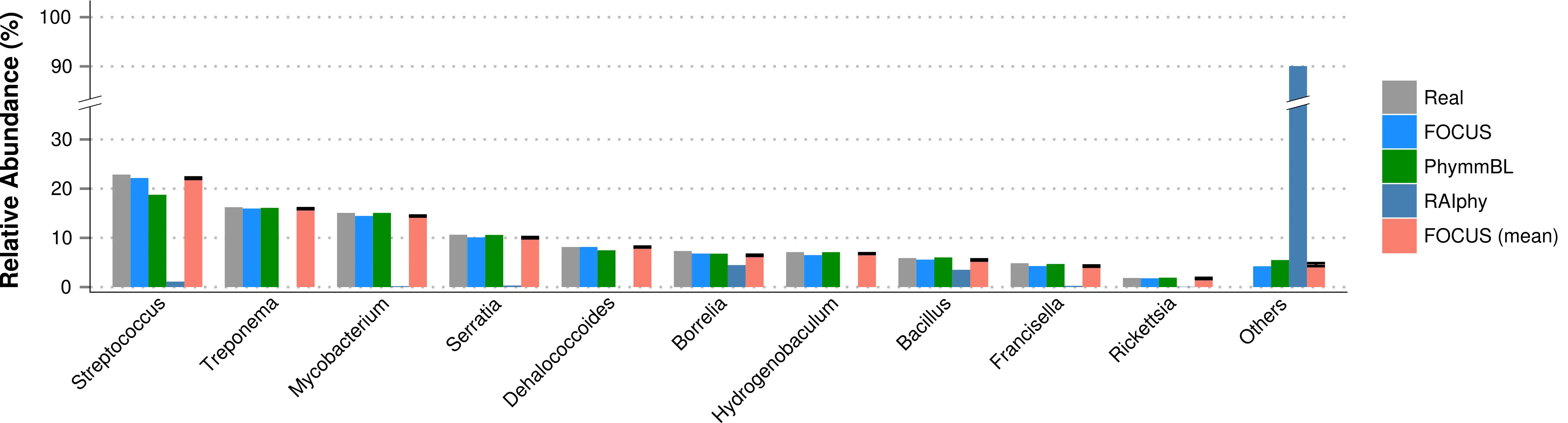
FOCUS, an innovative and agile composition based using non-negative least squares to profile and report abundant organisms present in me-tagenomic samples and their relative abundance without sequence length dependencies. The program was tested with simulated and real metagenomes, and the results show that our approach predicts the organisms in random communities.Availability and implementation: The code implemented in Python can be found here and a web-sever at http://edwards.sdsu.edu/FOCUS.
Dependencies: Jellyfish, Numpy, and Scipy.
Cite FOCUS
Silva, G. G. Z., D. A. Cuevas, B. E. Dutilh, and R. A. Edwards, 2014: FOCUS: an alignment-free model to identify organisms in metagenomes using non-negative least squares. PeerJ, 2, e425,doi:10.7717/peerj.425.
Features
- Metagenomes
- Richness
- Evenness
This is an application that can also be fetched from https://sourceforge.net/projects/metagenomefocus/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.