This is the Windows app named FineSplice whose latest release can be downloaded as FineSplice-0.2.2.tar.gz. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named FineSplice with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start any OS OnWorks online emulator from this website, but better Windows online emulator.
- 5. From the OnWorks Windows OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application and install it.
- 7. Download Wine from your Linux distributions software repositories. Once installed, you can then double-click the app to run them with Wine. You can also try PlayOnLinux, a fancy interface over Wine that will help you install popular Windows programs and games.
Wine is a way to run Windows software on Linux, but with no Windows required. Wine is an open-source Windows compatibility layer that can run Windows programs directly on any Linux desktop. Essentially, Wine is trying to re-implement enough of Windows from scratch so that it can run all those Windows applications without actually needing Windows.
SCREENSHOTS
Ad
FineSplice
DESCRIPTION
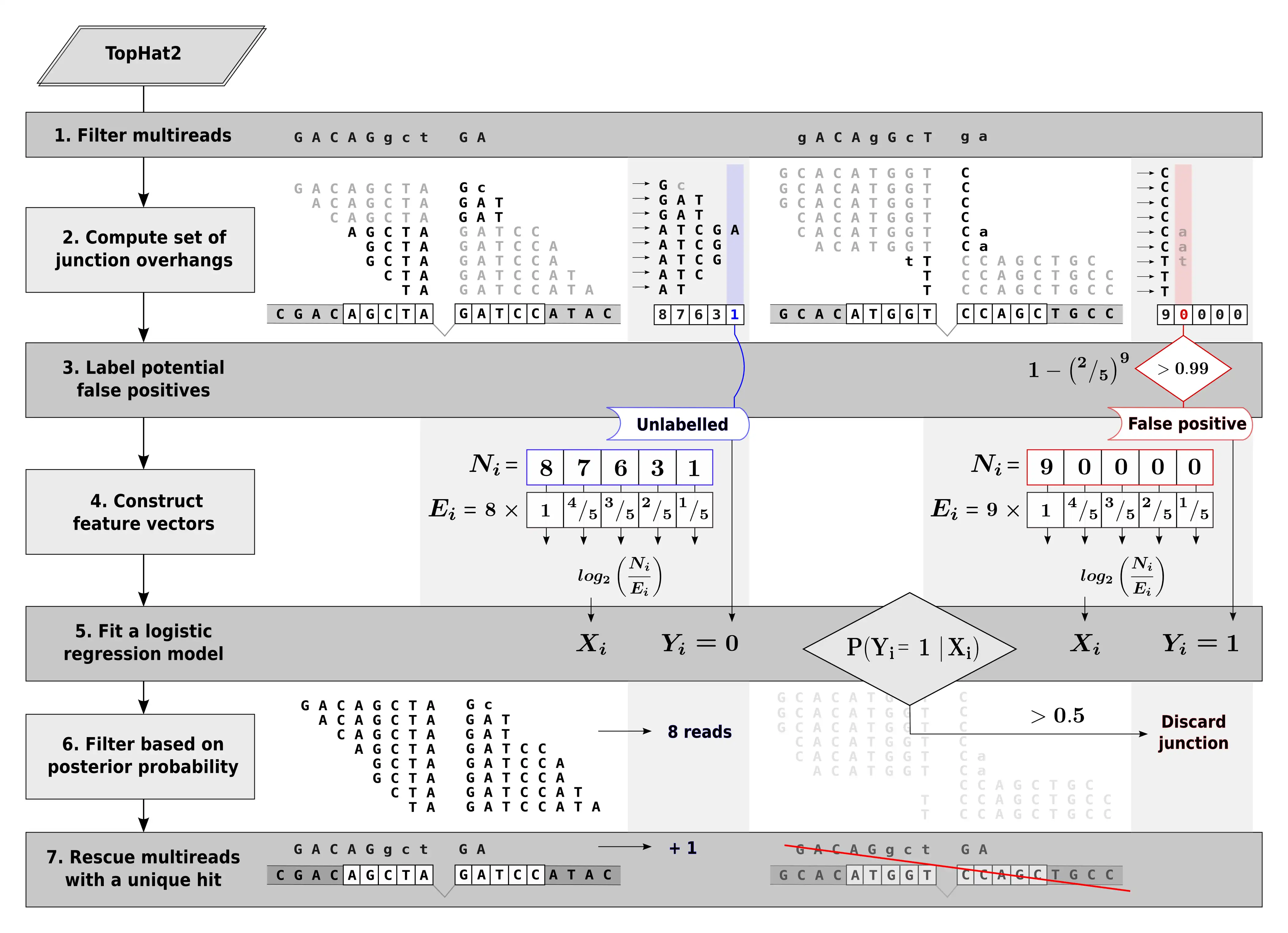
FineSplice is a Python wrapper to TopHat2 geared towards a reliable identification of expressed exon junctions from RNA-Seq data, at enhanced detection precision with small loss in sensitivity.
Following alignment with TopHat2 using known transcript annotations, FineSplice takes as input the resulting BAM file and outputs a confident set of expressed splice junctions with the corresponding read counts. Potential false positives arising from spurious alignments are filtered out via a semi-supervised anomaly detection strategy based on logistic regression. Multiple mapping reads with a unique location after filtering are rescued and reallocated to the most reliable candidate location.
FineSplice requires Python 2.x (>= 2.6) with the following modules installed: pysam (http://code.google.com/p/pysam/) and scikit-learn (http://scikit-learn.org/).
For further details check out our publication: Nucl. Acids Res. (2014) doi: 10.1093/nar/gku166
Features
- Align with TopHat2 using transcript annotations for superior mapping performance
- Run FineSplice to get rid of unreliable gapped alignments and improve splice junction detection precision
- Outputs a confident set of exon-exon junctions with the corresponding read counts for downstream analyses
Programming Language
Python
Categories
This is an application that can also be fetched from https://sourceforge.net/projects/finesplice/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.