This is the Linux app named locusvu to run in Linux online whose latest release can be downloaded as LocusVu.tar.gz. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named locusvu to run in Linux online with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start the OnWorks Linux online or Windows online emulator or MACOS online emulator from this website.
- 5. From the OnWorks Linux OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application, install it and run it.
SCREENSHOTS
Ad
locusvu to run in Linux online
DESCRIPTION
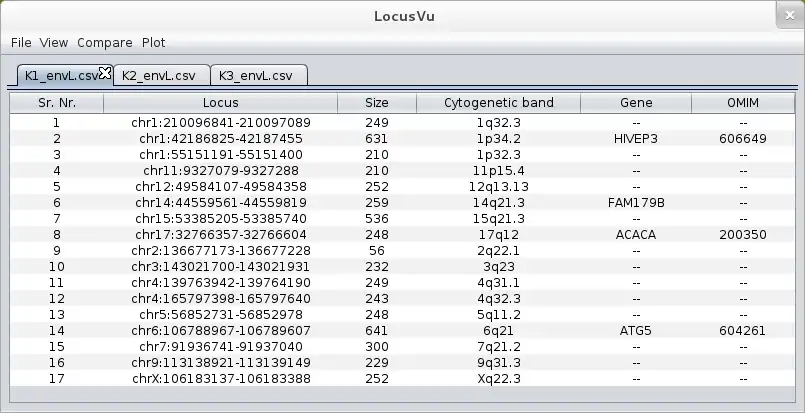
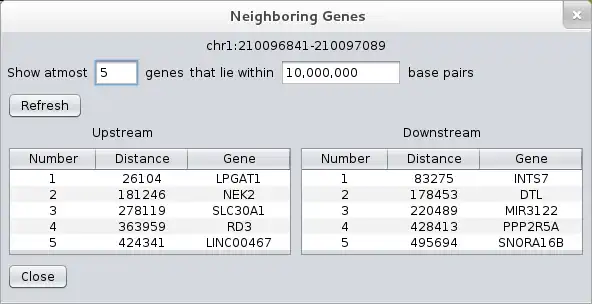
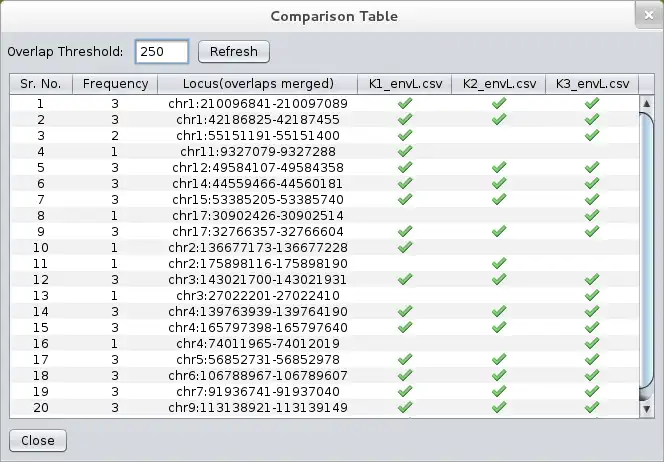
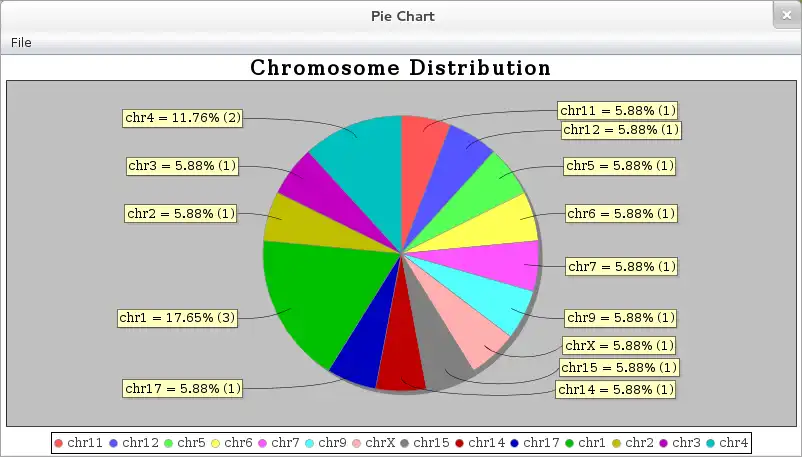
LocusVu is a novel Java based software tool that accepts a list of genomic loci (positions on the chromosome) as input and automates fetching of related information (cytogenetic band, gene name, OMIM data etc.) from public databases such as the UCSC genome browser database. It then enables multiple workflows on the retrieved results, like comparing multiple datasets (comparative genomics), viewing neighboring genes for a loci from within the tool itself, or graphically representing these results in bar / pie charts. LocusVu has a simple easy-to-use GUI on the frontend which enables intuitive user interaction with the underlying logic.The tool can be easily extended to add support for other databases (eg. Ensembl) or to fetch information
from other tables in the database. Thus, LocusVu provides a basic framework that can be used by users and developers alike in simplifying existing workflows and creating newer ones to support data analysis in genomics.
Features
- Automates data retrieval from databases (presently UCSC genome browser database)
- Enables workflows on retrieved results like..
- ..comparing multiple datasets (comparative genomics)
- ..view neighboring genes for a locus (from within the tool itself)
- ..graphically represent results (bar / pie charts)
- Easy-to-use GUI on the frontend for intuitive usage
- Logging with Log4j for easy debugging
- Easily extensible
Audience
Science/Research, Developers
User interface
Java Swing
Programming Language
Java
Database Environment
MySQL
This is an application that can also be fetched from https://sourceforge.net/projects/locusvu/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.