This is the Windows app named OpenGrowth to run in Windows online over Linux online whose latest release can be downloaded as OpenGrowth_Manual_1.0.pdf. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named OpenGrowth to run in Windows online over Linux online with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start any OS OnWorks online emulator from this website, but better Windows online emulator.
- 5. From the OnWorks Windows OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application and install it.
- 7. Download Wine from your Linux distributions software repositories. Once installed, you can then double-click the app to run them with Wine. You can also try PlayOnLinux, a fancy interface over Wine that will help you install popular Windows programs and games.
Wine is a way to run Windows software on Linux, but with no Windows required. Wine is an open-source Windows compatibility layer that can run Windows programs directly on any Linux desktop. Essentially, Wine is trying to re-implement enough of Windows from scratch so that it can run all those Windows applications without actually needing Windows.
SCREENSHOTS
Ad
OpenGrowth to run in Windows online over Linux online
DESCRIPTION
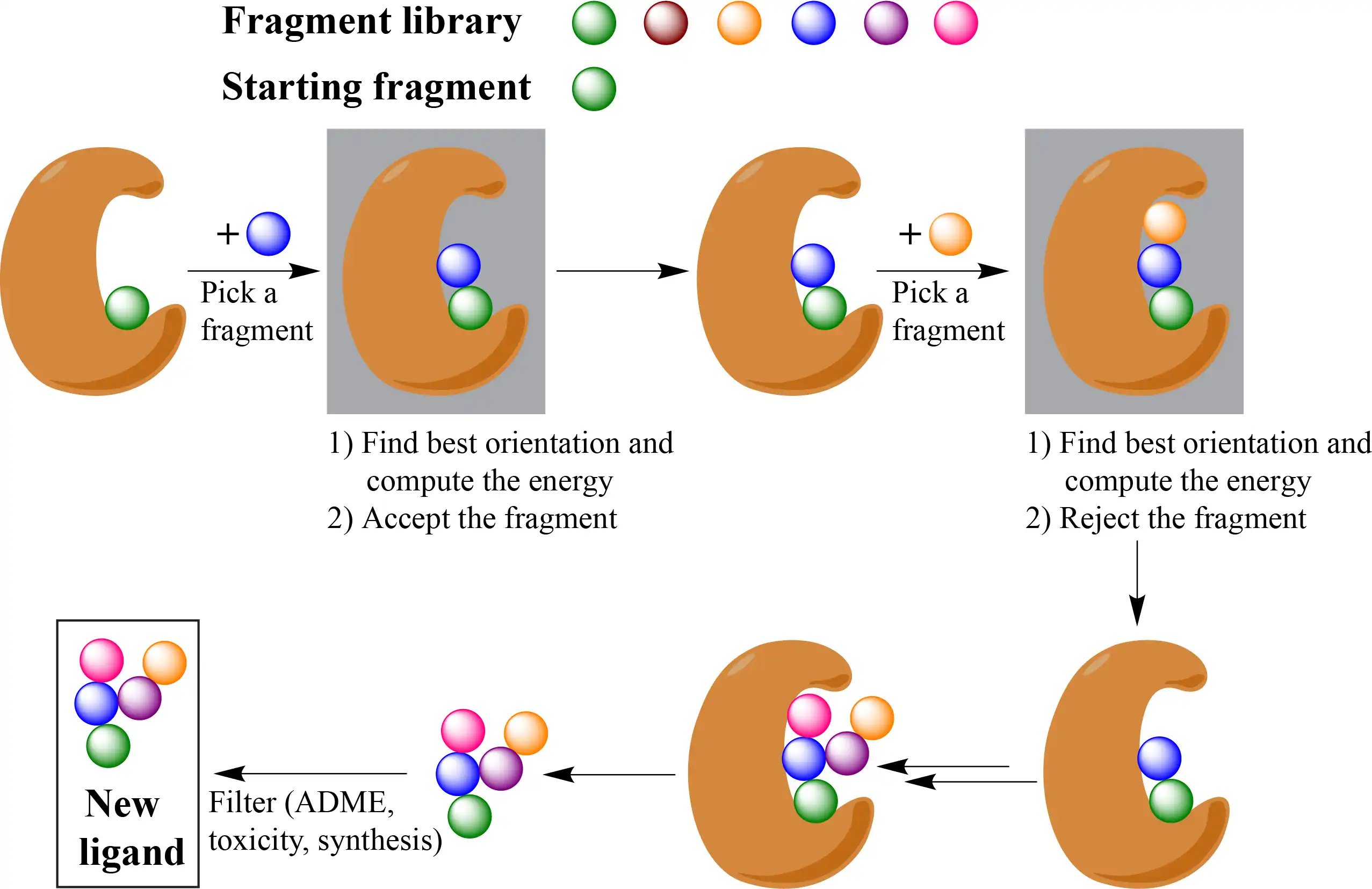
OpenGrowth is a research program which grows new ligands in proteins by connecting small organic fragments. The details can be found in the original publication "OpenGrowth: an automated and rational algorithm for finding new protein ligands" (J. Med. Chem., http://dx.doi.org/10.1021/acs.jmedchem.5b00886).To use OpenGrowth, you will need OpenGrowth_1.0.zip and Resources_1.0.2.zip that can be found by clicking on the Files menu in the horizontal bar at the top (https://sourceforge.net/projects/opengrowth/files). OpenGrowthGUI, FOG2.0, and the 3Mer-Screen stand-alone can be found in OpenGrowth_1.0.zip. To prepare new fragments you need BuildingFragments_1.0.1.zip and the scripts for MD simulations are in MD-Scripts_1.0.1.zip. SMoG2016.tar.gz allows to compute a score with the newly developed function (J. Chem. Inf. Mod., http://pubs.acs.org/doi/abs/10.1021/acs.jcim.6b00610).
For any questions, please send emails exclusively at [email protected].
Features
- Drug design
Audience
Healthcare Industry, Science/Research, End Users/Desktop
User interface
Qt
Programming Language
C++
This is an application that can also be fetched from https://sourceforge.net/projects/opengrowth/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.