This is the Linux app named lr2rmats to run in Linux online whose latest release can be downloaded as lr2rmats-v0.1-alpha.tar.gz. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named lr2rmats to run in Linux online with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start the OnWorks Linux online or Windows online emulator or MACOS online emulator from this website.
- 5. From the OnWorks Linux OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application, install it and run it.
SCREENSHOTS
Ad
lr2rmats to run in Linux online
DESCRIPTION
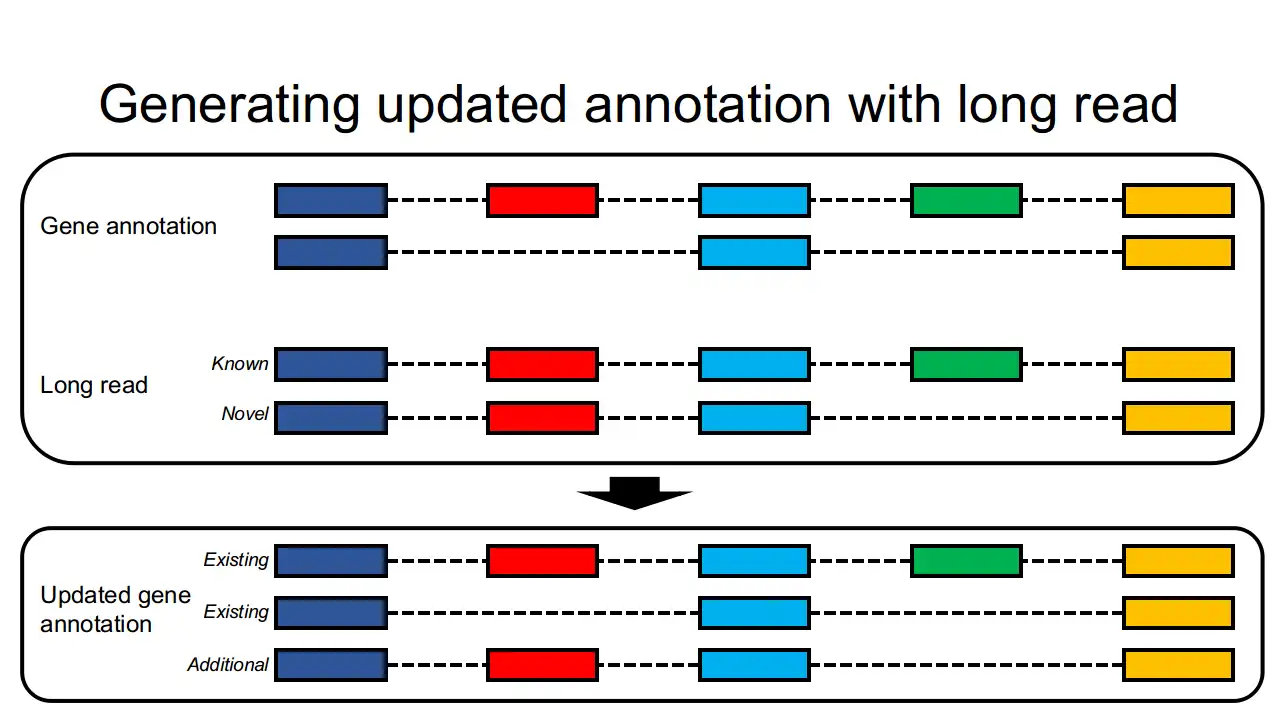
lr2rmats is a Snakemake-based light-weight pipeline which is designed to utilize both third-generation long-read and second-generation short-read RNA-seq data to generate an enhanced gene annotation file. The newly generated annotation file could be provided to rMATS for differential alternative splicing analysis.More information can be found at https://sourceforge.net/p/lr2rmats/wiki/Home/
Features
- Long read
- RNA-seq
- Alternative splicing
- Pacific Biosciences
- Oxford Nanopore Technologies
Audience
Science/Research
User interface
Command-line
Programming Language
Unix Shell, Python, C
This is an application that can also be fetched from https://sourceforge.net/projects/lr2rmats/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.