This is the Windows app named COV2HTML whose latest release can be downloaded as map2cov_4.4_WIN.zip. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named COV2HTML with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start any OS OnWorks online emulator from this website, but better Windows online emulator.
- 5. From the OnWorks Windows OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application and install it.
- 7. Download Wine from your Linux distributions software repositories. Once installed, you can then double-click the app to run them with Wine. You can also try PlayOnLinux, a fancy interface over Wine that will help you install popular Windows programs and games.
Wine is a way to run Windows software on Linux, but with no Windows required. Wine is an open-source Windows compatibility layer that can run Windows programs directly on any Linux desktop. Essentially, Wine is trying to re-implement enough of Windows from scratch so that it can run all those Windows applications without actually needing Windows.
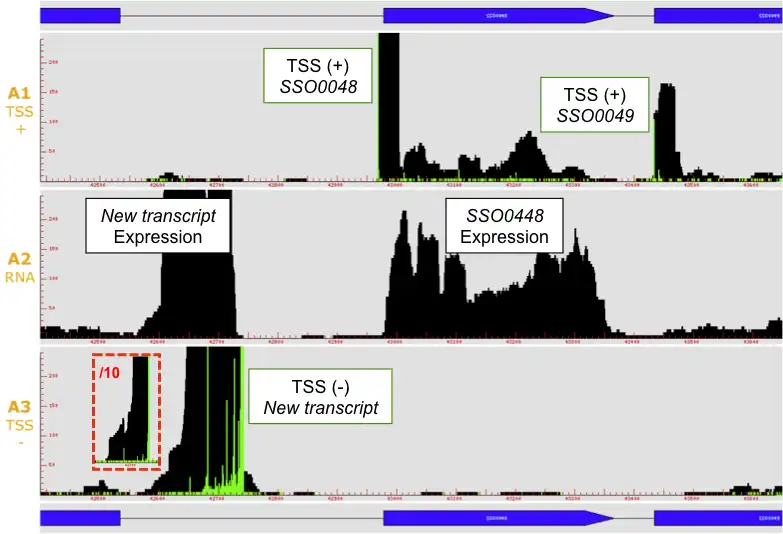
SCREENSHOTS
Ad
COV2HTML
DESCRIPTION
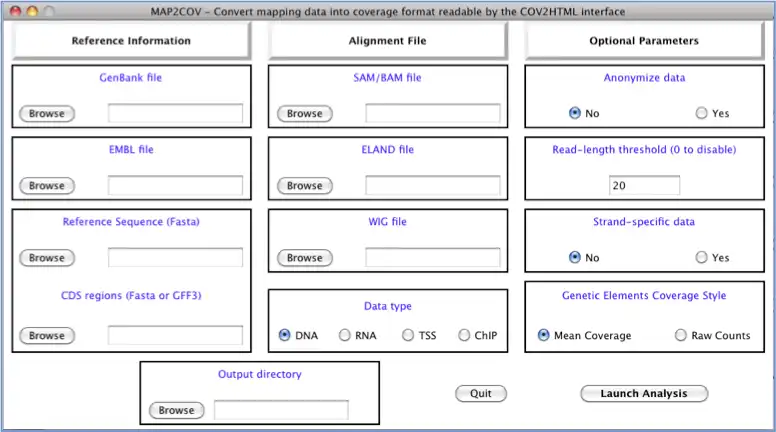
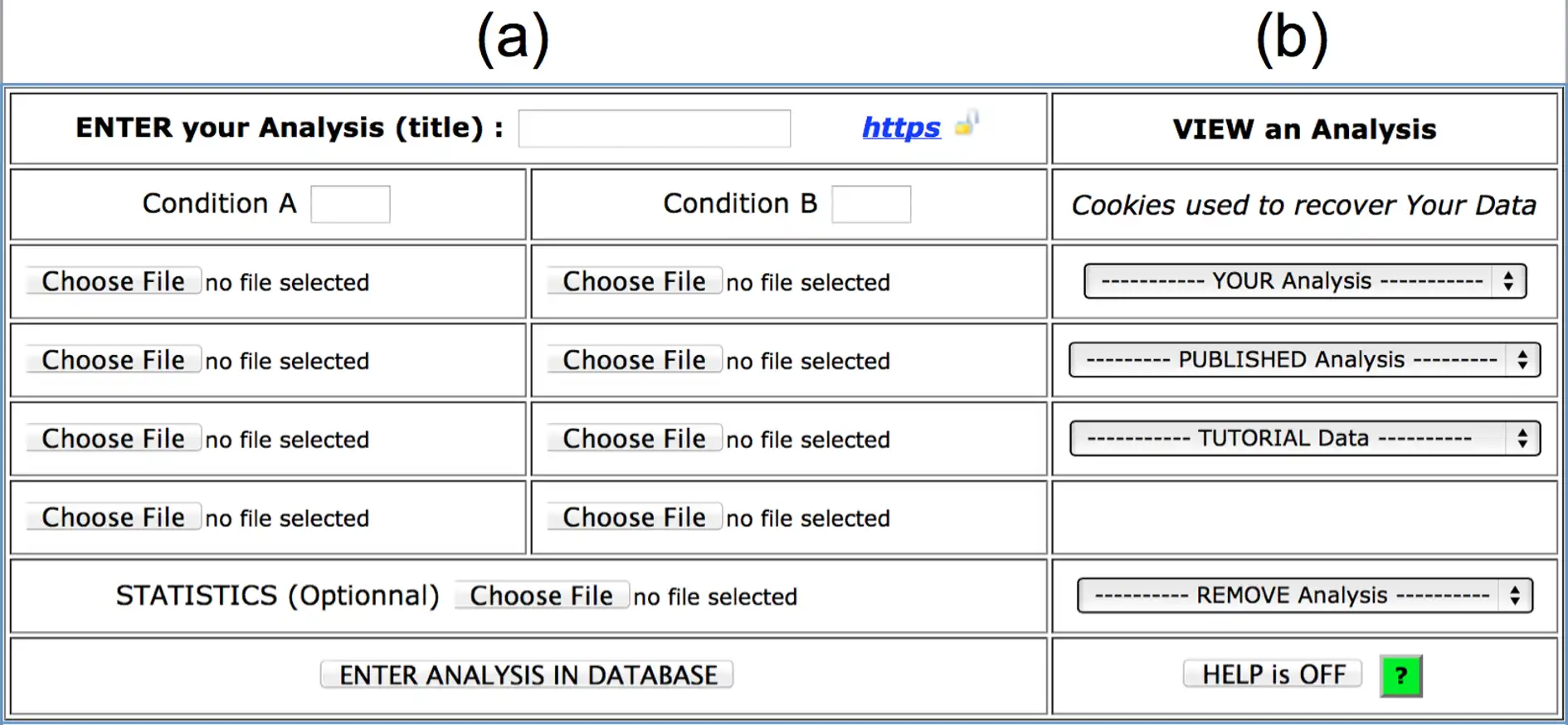
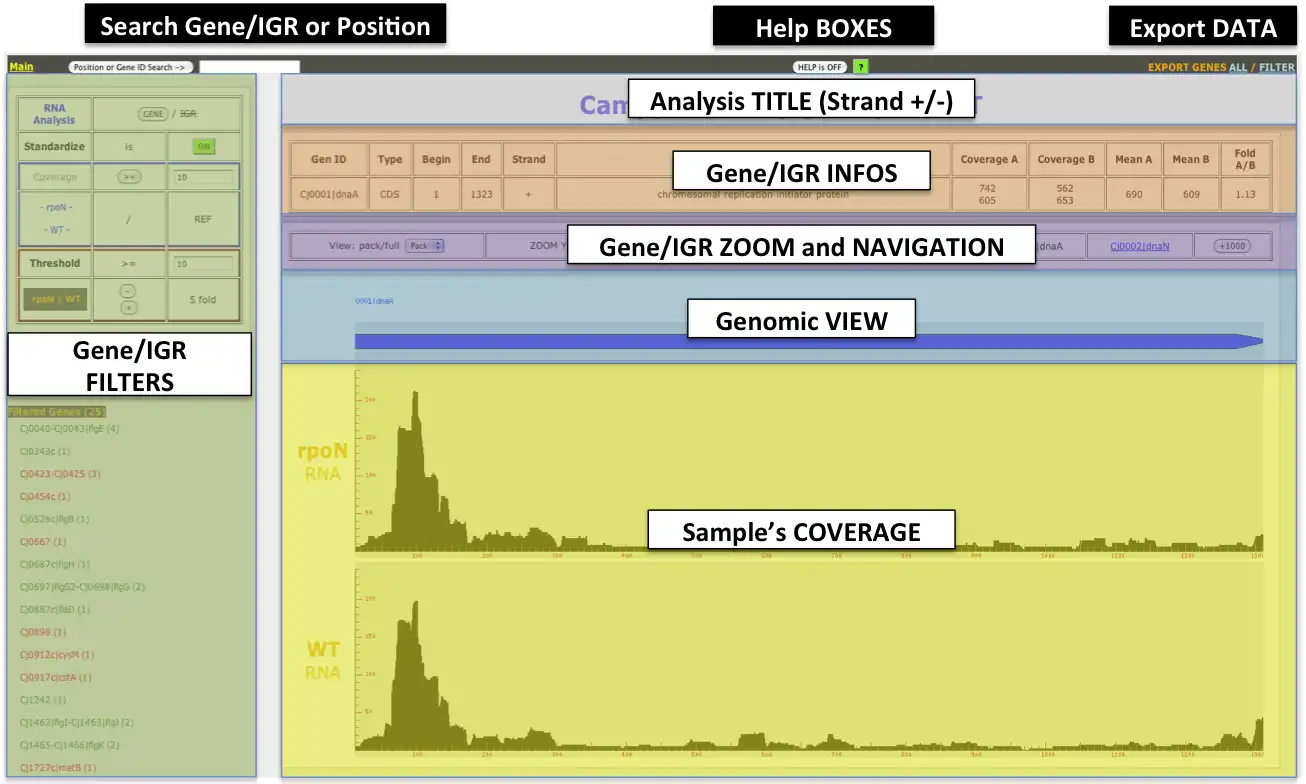
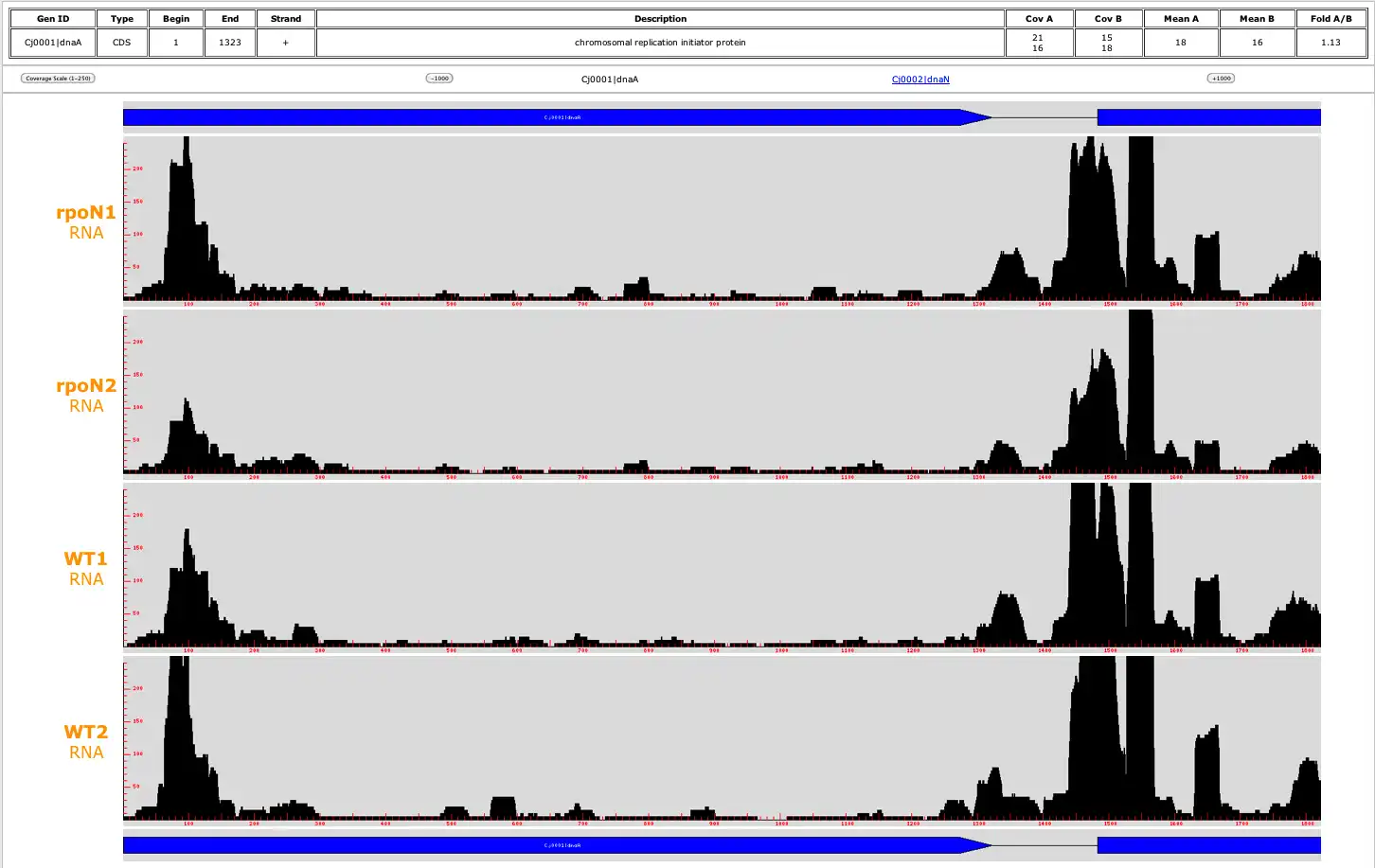
COV2HTML provides an easy and 'in home' web interface for biologists that allows coverage visualization of the NGS alignment needed for the analysis. It combines two essential processes: (i) MAP2COV, a tool that converts the huge NGS mapping or coverage files into light specific coverage files which contains genetic elements informations. (ii) COV2HTML, a visualization interface allowing a real-time analysis of data with selected criteria. Thus this interface offers a visualization of NGS mapping coverage data (DNA-seq, RNA-seq, ChIP-seq and TSS) performed in different prokaryotic organisms (bacteria, viruses...) or different experimental conditions (mutant versus wild type strains or different growth states…) facilitating studies.
Features
- RNA-Seq strand coverage visualization : Black (strand +), Purple (strand -)
- ncRNA feature analysis
- X and Y Zooms are supported
- Security enhanced: HTTPS as default
Audience
Science/Research
User interface
Project is a user interface (UI) system, Tk
Programming Language
Python, PHP
Database Environment
MySQL
Categories
This is an application that can also be fetched from https://sourceforge.net/projects/cov2html/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.