This is the Linux app named Kinannote to run in Linux online whose latest release can be downloaded as Kinannote_1.0.tar. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named Kinannote to run in Linux online with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start the OnWorks Linux online or Windows online emulator or MACOS online emulator from this website.
- 5. From the OnWorks Linux OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application, install it and run it.
SCREENSHOTS
Ad
Kinannote to run in Linux online
DESCRIPTION
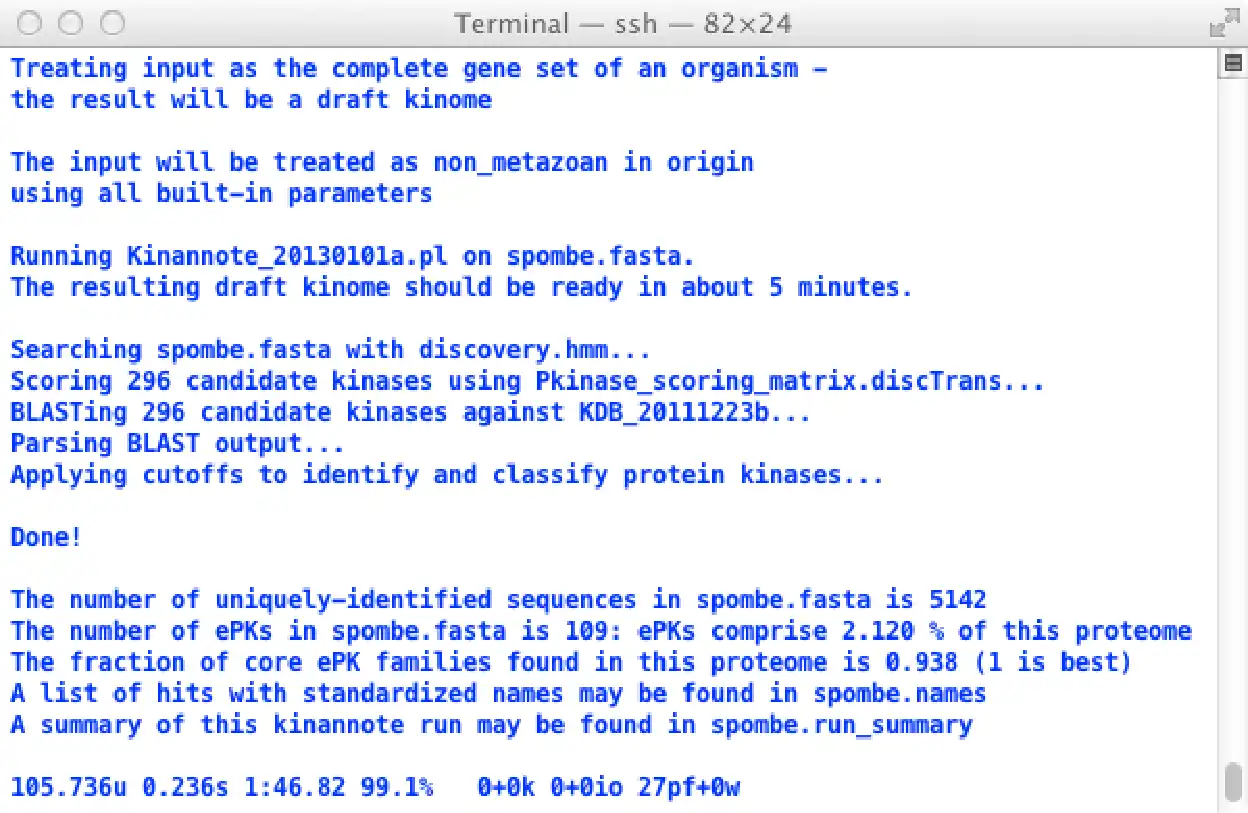
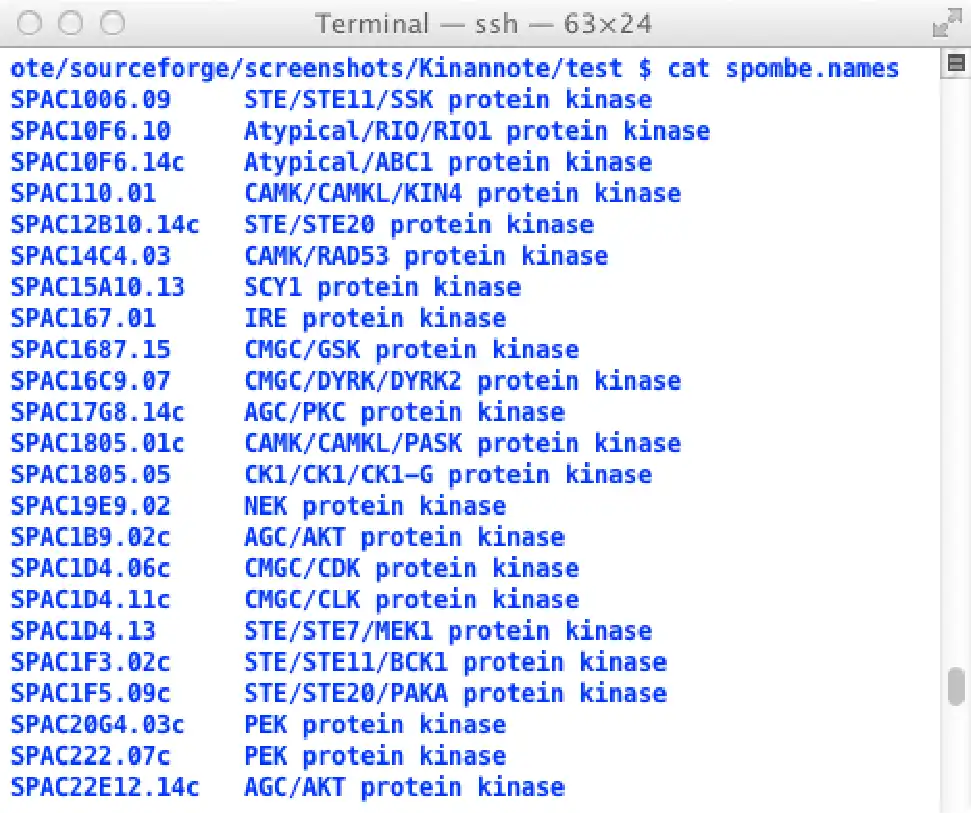
Kinannote identifies and classifies protein kinases in a user-provided fasta file using an HMM derived from serine/threonine protein kinases, a position specific scoring matrix derived from the HMM, and comparison with a local version of the curated kinase database from kinase.com. If the user inputs a complete proteome, additional modules evaluate the completeness of the kinome and place it in context with reference kinomes. Kinannote runs on a unix command line and depends on local hmmer 2 and Blast 2.24 installations.Citing Kinannote:
Kinannote, a computer program to identify and classify members of the eukaryotic protein kinase superfamily
Jonathan M. Goldberg; Allison Griggs; Janet L. Smith; Brian Haas; Jennifer Wortman; Qiandong Zeng
Bioinformatics 2013; doi: 10.1093/bioinformatics/btt419
http: http://bioinformatics.oxfordjournals.org/content/29/19/2387.full
pdf: http://bioinformatics.oxfordjournals.org/content/29/19/2387.full.pdf+html
Audience
Science/Research
Programming Language
Perl
This is an application that can also be fetched from https://sourceforge.net/projects/kinannote/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.