This is the Linux app named HomSI to run in Linux online whose latest release can be downloaded as HomSI.zip. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named HomSI to run in Linux online with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start the OnWorks Linux online or Windows online emulator or MACOS online emulator from this website.
- 5. From the OnWorks Linux OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application, install it and run it.
SCREENSHOTS
Ad
HomSI to run in Linux online
DESCRIPTION
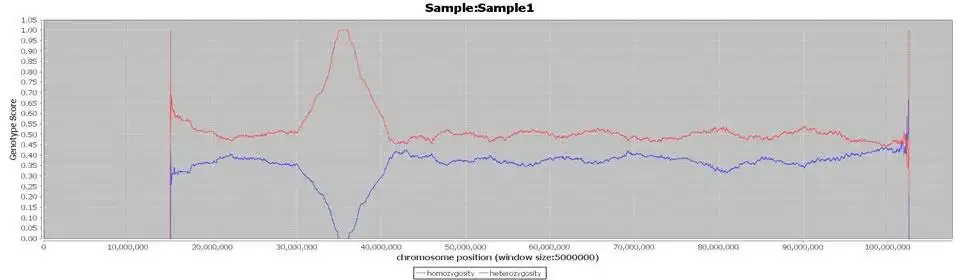
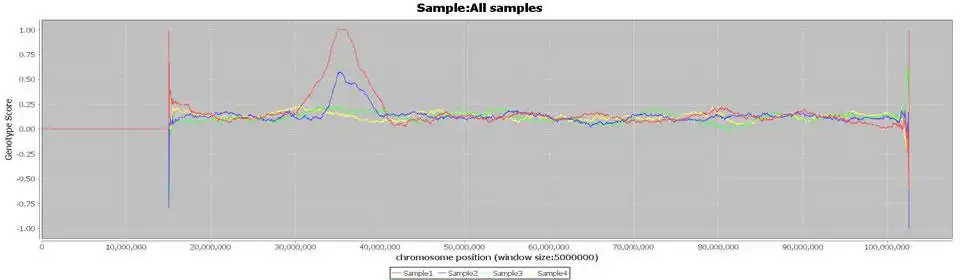
In consanguineous families, as a result of inheriting the same genomic segments through both parents, the individuals have stretches of their genomes that are homozygous. This situation leads to the prevalence of recessive diseases among the members of these families. Homozygosity mapping is based on this observation and several recessive disease genes have been discovered with the help of this technique in consanguineous families. The researchers typically use SNP arrays to determine the homozygous regions and then search for the disease gene by sequencing the genes within this candidate disease loci. Recently, the advent of next generation sequencing enables the concurrent identification of homozygous regions and the detection of mutations relevant for diagnosis, using data from a single sequencing experiment. In this respect, we have developed a novel tool that identifies homozygous regions using deep sequence data. Using *.vcf files as an input file, our program identifies the majoFeatures
- Homozygosity Mapping on NGS data
User interface
Java SWT
Programming Language
Java
This is an application that can also be fetched from https://sourceforge.net/projects/homsi/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.