This is the Linux app named LymPHOS2 whose latest release can be downloaded as LymPHOS2Presentation2017-05-21v0.9.9.odp. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named LymPHOS2 with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start the OnWorks Linux online or Windows online emulator or MACOS online emulator from this website.
- 5. From the OnWorks Linux OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application, install it and run it.
SCREENSHOTS
Ad
LymPHOS2
DESCRIPTION
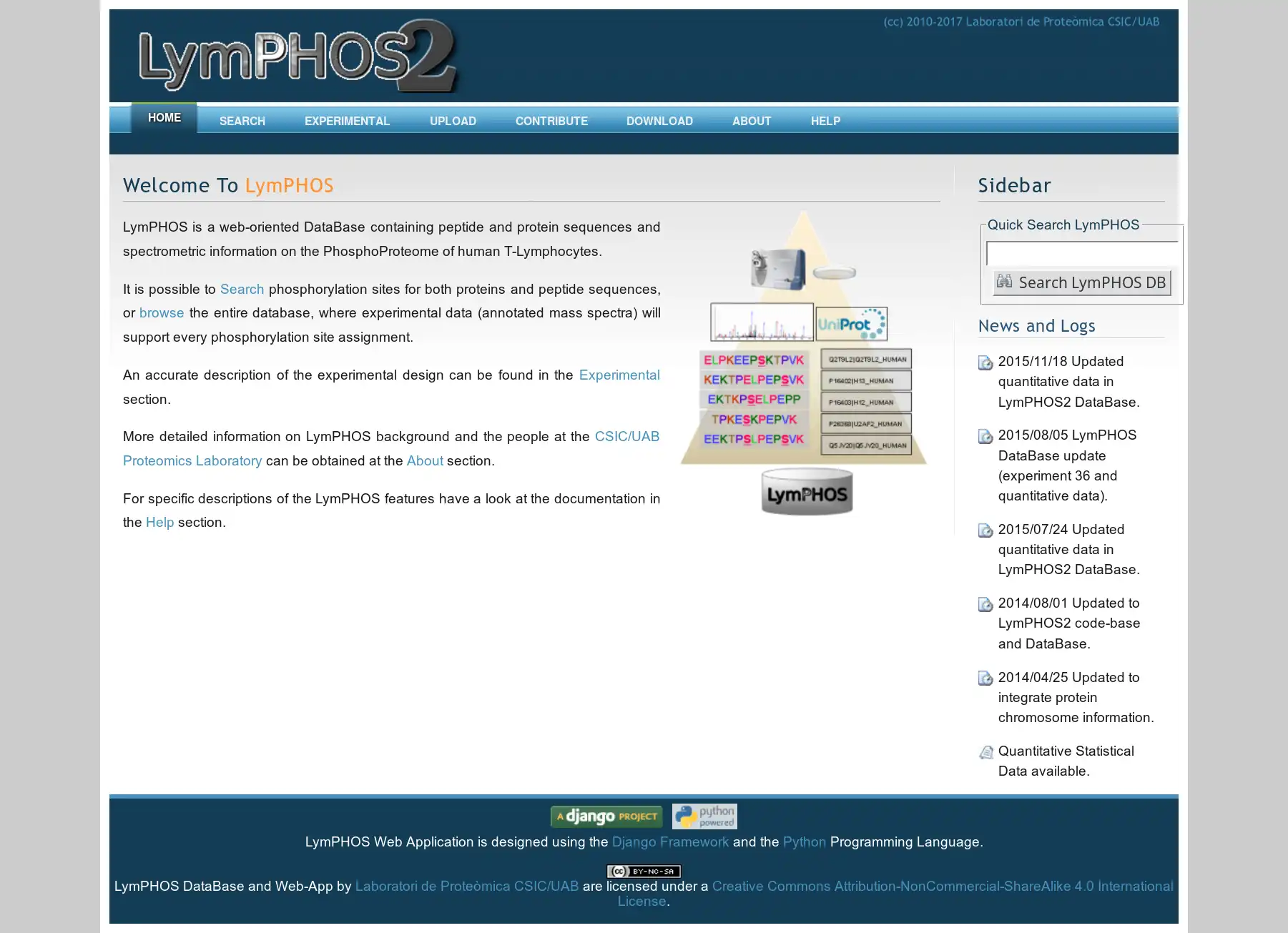
LymPHOS2 is a web-based Application at www.LymPHOS.org containing peptidic and protein sequences and spectrometric information on the PhosphoProteome of human T-Lymphocytes.
- Nguyen, TD., Vidal-Cortes, O., Gallardo, Ó., Abian, J., Carrascal, M., LymPHOS 2.0: an update of a phosphosite database of primary human T cells. Database 2015, 2015. DOI: 10.1093/database/bav115
- Carrascal, M., Ovelleiro, D., Casas, V., Gay, M., Abian, J., Phosphorylation analysis of primary human T lymphocytes using sequential IMAC and titanium oxide enrichment. J. Proteome Res. 2008, 7, 5167-5176. DOI: 10.1021/pr800500r
- Ovelleiro, D., Carrascal, M., Casas, V., Abian, J., LymPHOS: design of a phosphosite database of primary human T cells. Proteomics 2009, 9, 3741–3751. DOI: 10.1002/pmic.200800701
- Gallardo, Ó., Ovelleiro, D., Gay, M., Carrascal, M., Abian, J., A collection of open source applications for mass spectrometry data mining. Proteomics 2014, 20, 2275-2279. DOI: 10.1002/pmic.20140012
Features
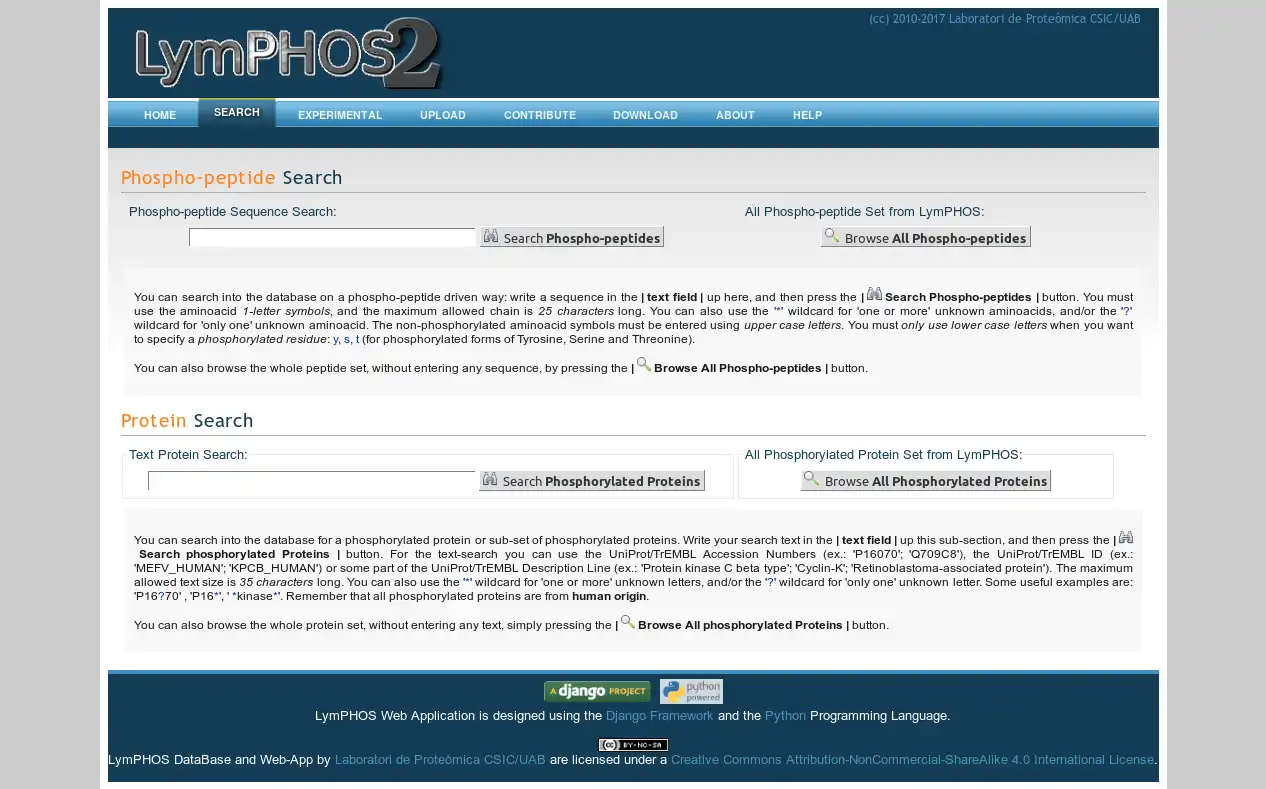
- On-line at https://www.LymPHOS.org/
- PhosphoProteomics Information for human T cells
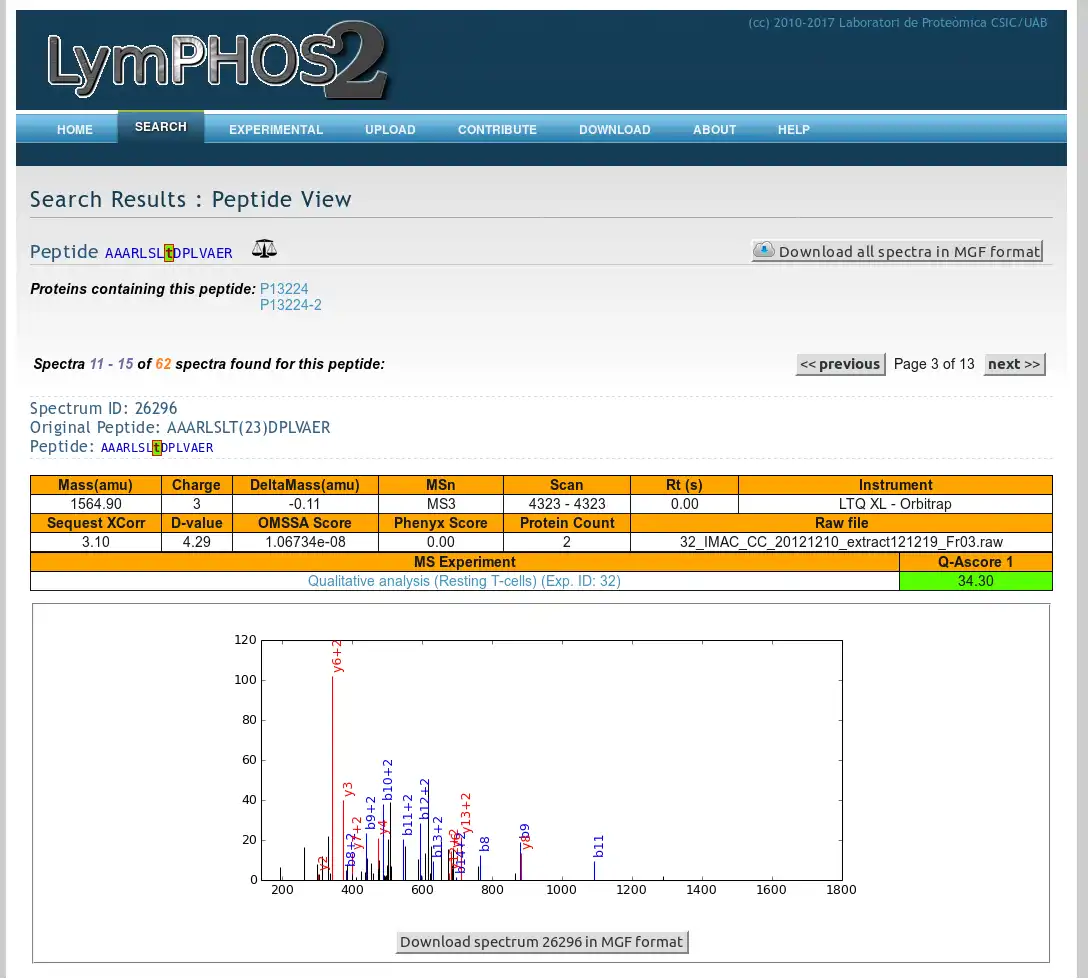
- Mass Spectra
- Proteomics Data
- Python-Django Web-App
- MySQL Database
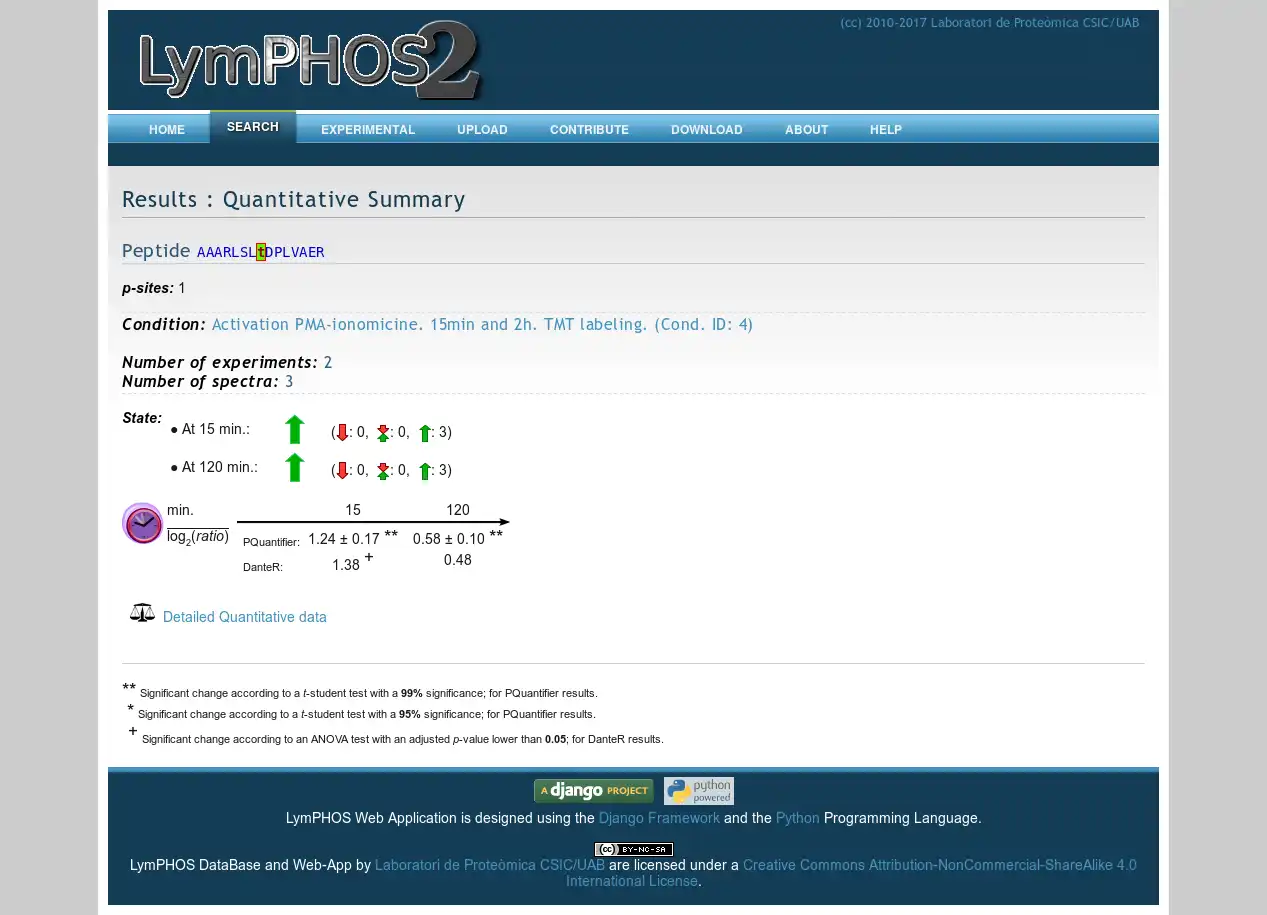
- PQuantifier quantification tool
- Conditions tool to enter experimental metadata
- Other analysis tools
- Extra Documentation
Audience
Science/Research
User interface
Web-based
Programming Language
Python
Database Environment
MySQL
Categories
This is an application that can also be fetched from https://sourceforge.net/projects/lymphosweb.lp-csic-uab.p/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.