This is the Linux app named MoPAC whose latest release can be downloaded as MoPAC_0.3.1.tar.gz. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named MoPAC with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start the OnWorks Linux online or Windows online emulator or MACOS online emulator from this website.
- 5. From the OnWorks Linux OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application, install it and run it.
SCREENSHOTS
Ad
MoPAC
DESCRIPTION
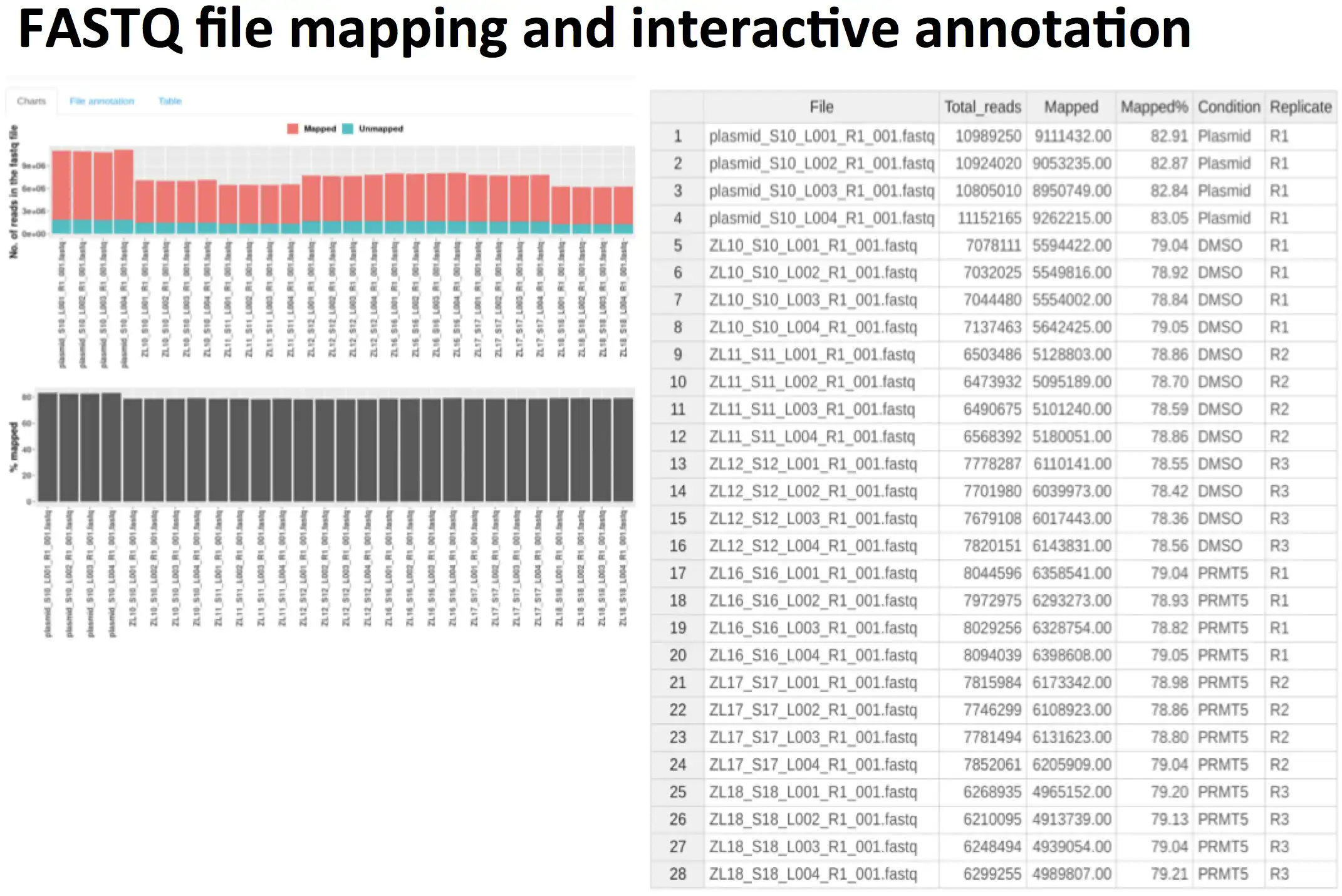
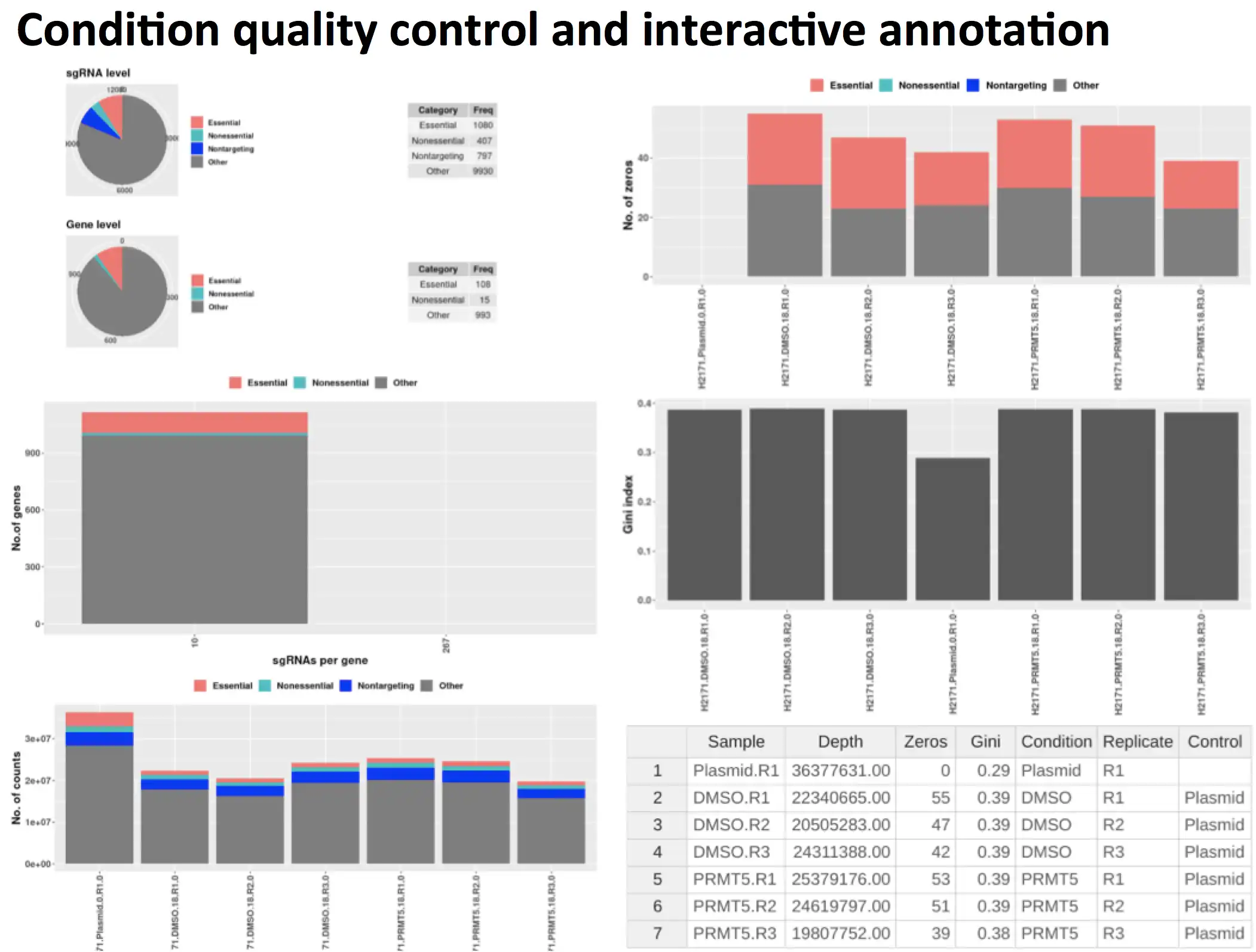
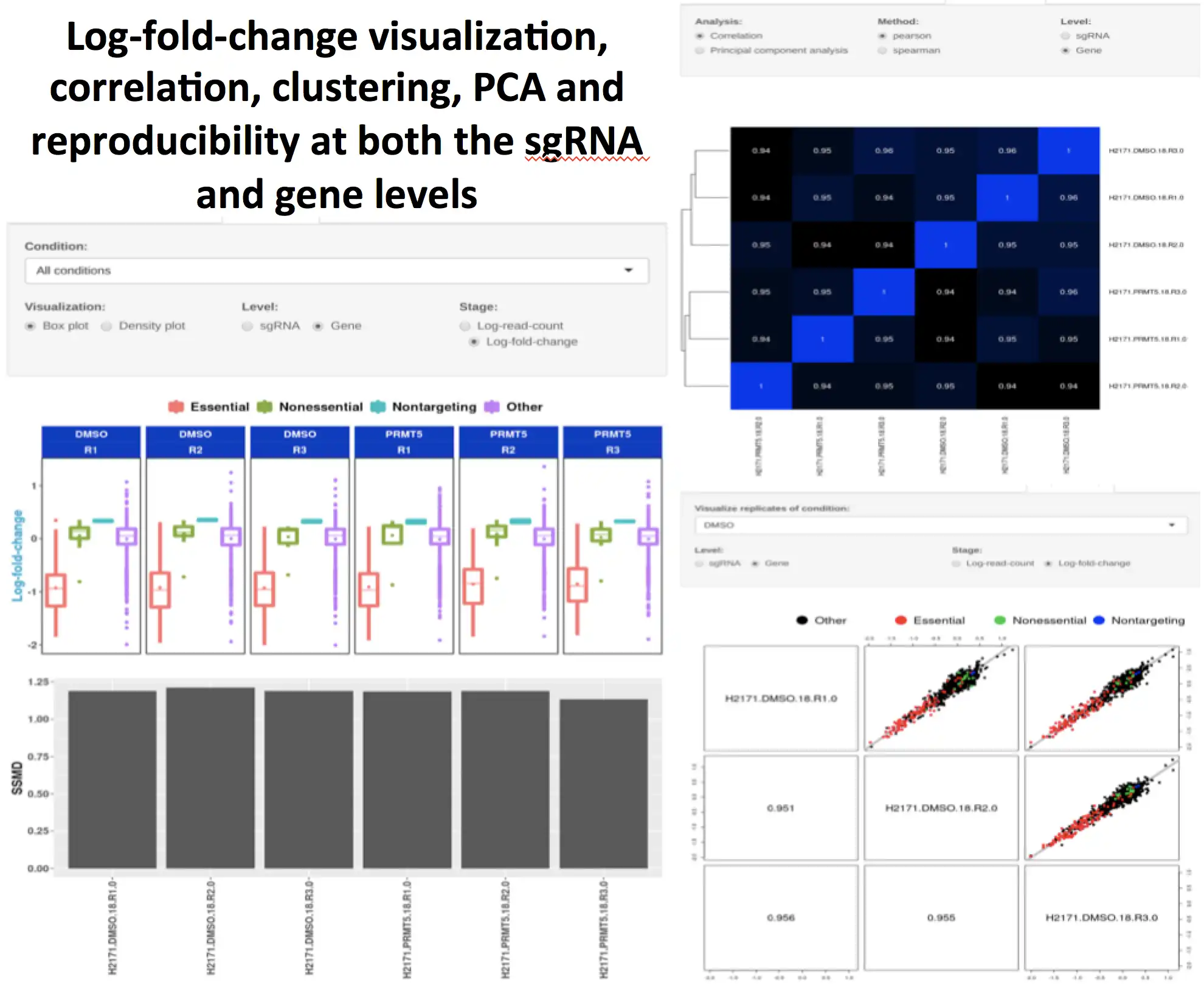
To facilitate the comparison of gene essentialities in two or more cell samples, we propose MoPAC (Modular Pipeline for Analysis of CRISPR screens), a Shiny-driven interactive tool for differential essentiality analysis in CRISPR/Cas9 screens.
For installation and usage instructions please refer to the wiki page.
Features
- Unbiased measurement of differential gene essentiality in CRISPR screens.
- Ease of use through an interactive graphical user interphase.
- Publication-ready figures generated at every step of the analysis.
- Both FASTQ files and read-count tables (before or after normalization) allowed as input.
- Gene-category-based quality control for outlier detection and filtering.
- Gene ontology analysis built-in through the STRINGdb R package.
- Fast processing through C++ compilation of calculation-intensive steps.
Audience
Science/Research
User interface
Web-based
Programming Language
C++, S/R
Categories
This is an application that can also be fetched from https://sourceforge.net/projects/mopac/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.