This is the Linux app named RNAseqR to run in Linux online whose latest release can be downloaded as RNAseqR_1.1.tar.gz. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named RNAseqR to run in Linux online with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start the OnWorks Linux online or Windows online emulator or MACOS online emulator from this website.
- 5. From the OnWorks Linux OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application, install it and run it.
SCREENSHOTS
Ad
RNAseqR to run in Linux online
DESCRIPTION
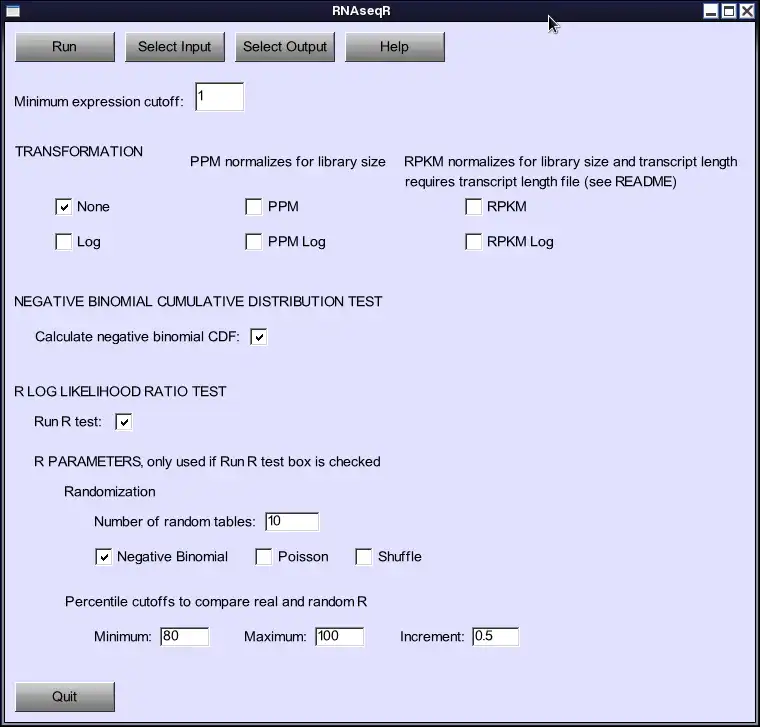
RNAseqR is designed for expression analysis of RNA-seq data, and is best suited for analysis of multiple, unreplicated libraries. It is a C++ coded program currently compiled for Linux systems.With GUI and command line interfaces, it can do log, PPM, and/or RPKM (if lengths provided) transformations, as well as, statistical analysis for differential expression, using the negative binomial cumulative distribution function (CDF) or the R test statistic introduced for EST analysis by Stekel, et al.
Output allows for decisions based on CDF probabilities, top R values, or comparison of R values with randomized data. Comparisons are the believability metric of Stekel et al, or observed vs expected R at a given mean expression. Randomization is through Poisson or negative binomial distributions, or column shuffling.
Programming Language
C++
This is an application that can also be fetched from https://sourceforge.net/projects/rnaseqr/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.