This is the Linux app named sgRNAcas9 whose latest release can be downloaded as sgRNAcas9_3.0.5.zip. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named sgRNAcas9 with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start the OnWorks Linux online or Windows online emulator or MACOS online emulator from this website.
- 5. From the OnWorks Linux OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application, install it and run it.
SCREENSHOTS
Ad
sgRNAcas9
DESCRIPTION
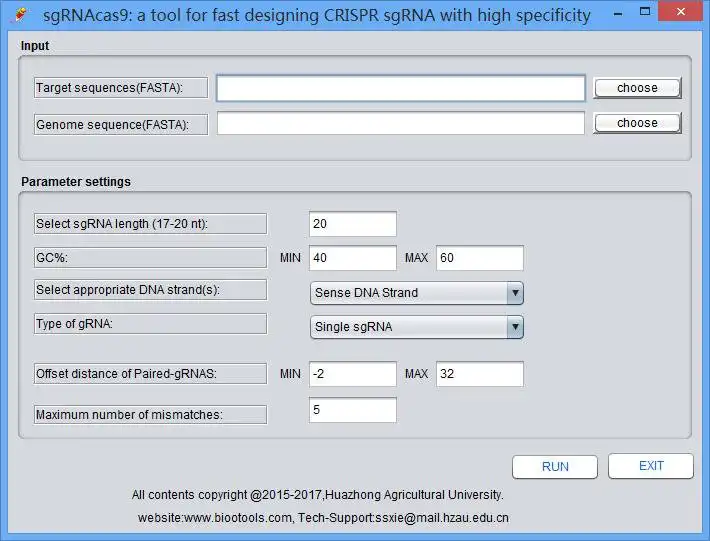
This package consists of programs to perform a search for CRISPR target sites (protospacers) with user-defined parameters, predict genome-wide Cas9 potential off-target cleavage sites (POT), classify the POT into three categories, batch design oligonucleotides for constructing 20-nt (nucleotides) or truncated sgRNA expression vectors, extract desired length nucleotide sequences flanking the on- or off-target cleavage sites for designing PCR primer pairs to validate the mutations by T7E1 cleavage assay. Importantly, by identifying potential off-target sites in silico, the sgRNAcas9 allows the selection of more specific target sites and aids the identification of bona fide off-target sites, significantly facilitating the design of sgRNA for genome editing applications.Cites: Xie S, ShenB,Zhang C, Huang X, Zhang Y. sgRNAcas9: a software package for designing CRISPR sgRNA and evaluating potential off-target cleavage sites. PLoS One. 2014,9(6):e100448.
http://www.biootools.com/
This is an application that can also be fetched from https://sourceforge.net/projects/sgrnacas9/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.