This is the Windows app named gsasnp2 to run in Windows online over Linux online whose latest release can be downloaded as gsasnp2-linux-cmd-ubuntu.zip. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named gsasnp2 to run in Windows online over Linux online with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start any OS OnWorks online emulator from this website, but better Windows online emulator.
- 5. From the OnWorks Windows OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application and install it.
- 7. Download Wine from your Linux distributions software repositories. Once installed, you can then double-click the app to run them with Wine. You can also try PlayOnLinux, a fancy interface over Wine that will help you install popular Windows programs and games.
Wine is a way to run Windows software on Linux, but with no Windows required. Wine is an open-source Windows compatibility layer that can run Windows programs directly on any Linux desktop. Essentially, Wine is trying to re-implement enough of Windows from scratch so that it can run all those Windows applications without actually needing Windows.
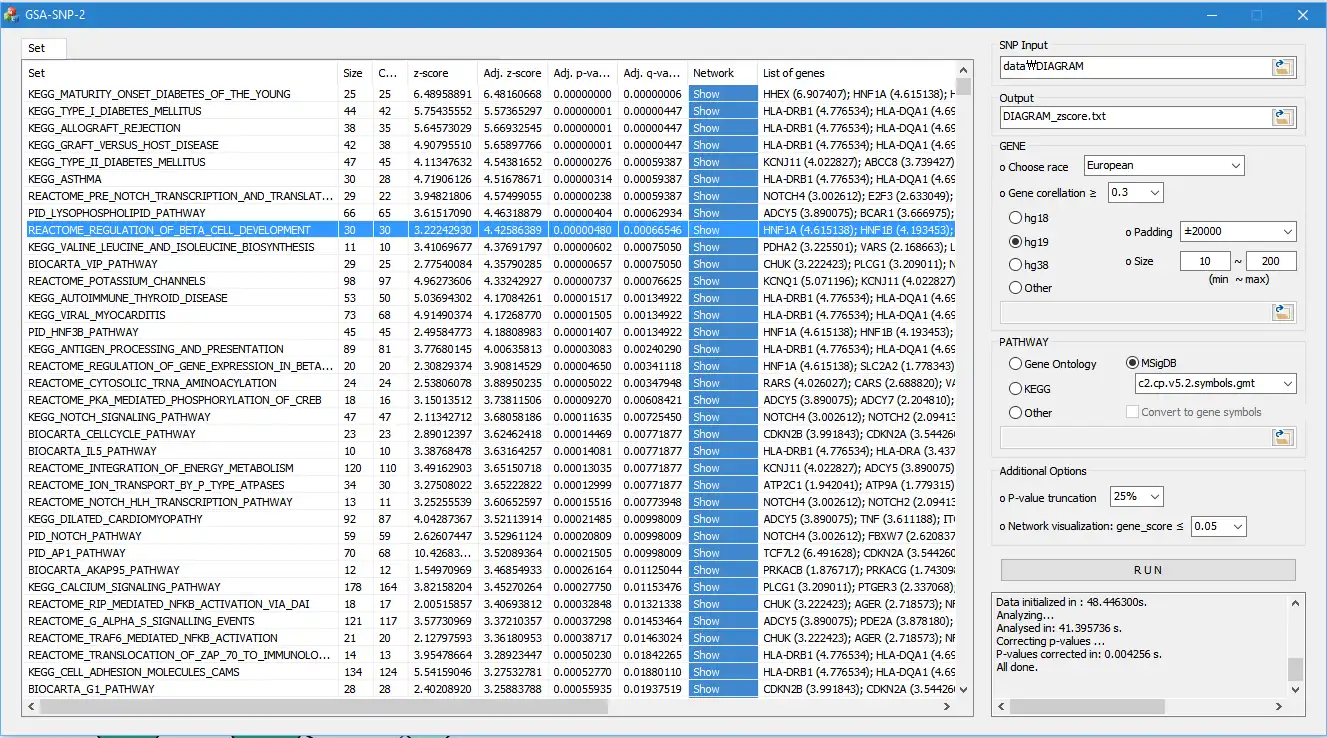
SCREENSHOTS
Ad
gsasnp2 to run in Windows online over Linux online
DESCRIPTION
* GSA-SNP2 is a successor of GSA-SNP (Nam et al. 2010, NAR web server issue). GSA-SNP2 accepts human GWAS summary data (rs numbers, p-values) or gene-wise p-values and outputs pathway genesets ‘enriched’ with genes associated with the given phenotype. It also provides both local and global protein interaction networks in the associated pathways.* Article: SYoon, HCTNguyen, YJYoo, JKim, BBaik, SKim, JKim, SKim, DNam, "Efficient pathway enrichment and network analysis of GWAS summary data using GSA-SNP2", Nucleic Acids Research, Vol. 46(10), e60(2018).
* PubMed ID: 29562348
* DOI: 10.1093/nar/gky175
-> PLEASE MOVE OR MAKE A COPY OF 'DATA' FOLDER INTO YOUR INTENSIVE TEST FOLDER (I.E. LINUX, MAC OR WINDOWS SPECIFIED FOLDER) TO ALLOW THE PROGRAM TO FIND THE PREDESIGNED DATA.
* UPDATE NOTE:
-> Aug-7-2019: add an update for Ubuntu-19.04. You will need Boost library installed (sudo apt-get install libboost-all-dev)
-> Mar-7-2018: revise header terms in the output file
Features
- 1/ 'DECENT TYPE I ERROR CONTROL' achieved by the following two processes: A) Gene scores are ‘adjusted’ to the number of SNPs assigned to each gene using monotone cubic spline trend curve. B) Adjacent genes with high inter-gene correlations within each pathway were removed
- 2/ 'HIGH POWER AND FAST COMPUTATION' based on the random set model
- 3/ 'NO CRITICAL FREE PARAMETER'
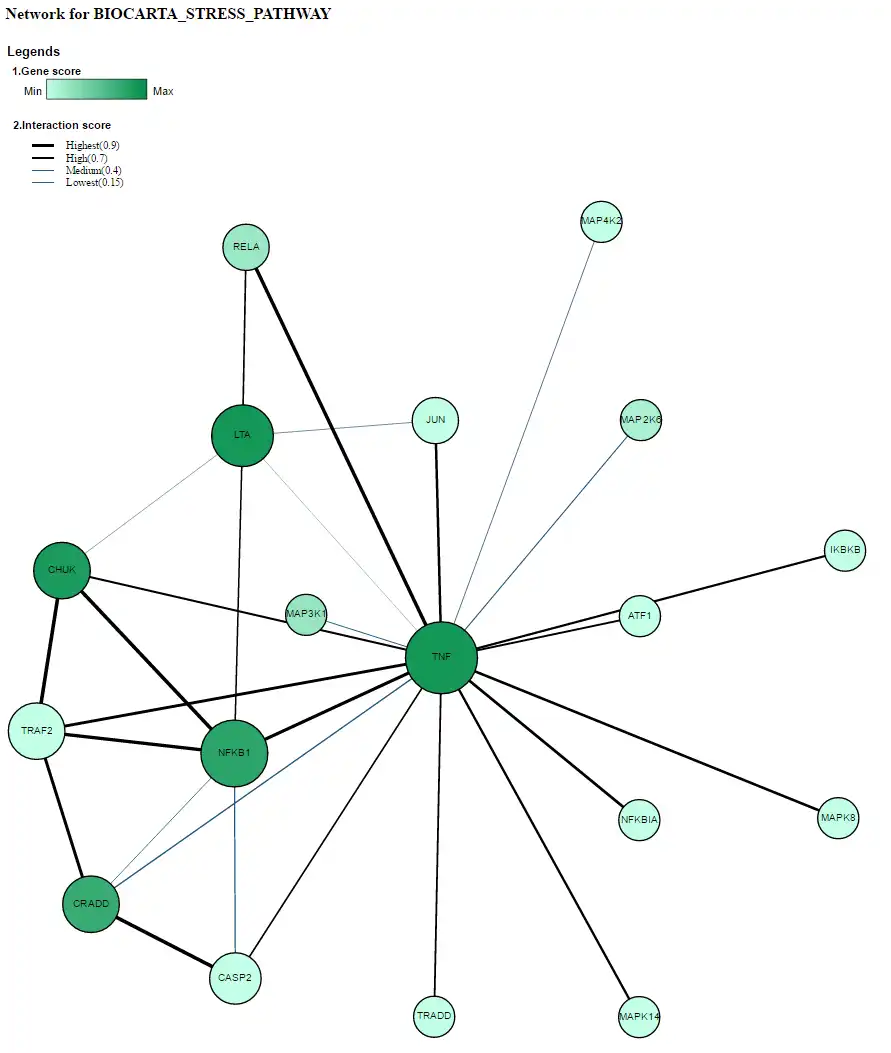
- 4/ 'PROTEIN INTERACTION NETWORKS' among the member genes were visualized for the significant pathways. This function enables the user to prioritize the core sub-networks within and across the significant pathways. The STRING and HIPPIE networks are currently provided
- 5/ 'EASY TO USE': It only requires GWAS summary data (or gene p-values) and takes only a minute or two to get results. Other powerful self-contained pathway tools require the SNP correlation input as well and take a much longer time. User can also upload their own pathway gene-sets and protein interaction networks.
User interface
Win32 (MS Windows), Command-line
Programming Language
C++, PHP, JavaScript
This is an application that can also be fetched from https://sourceforge.net/projects/gsasnp2/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.